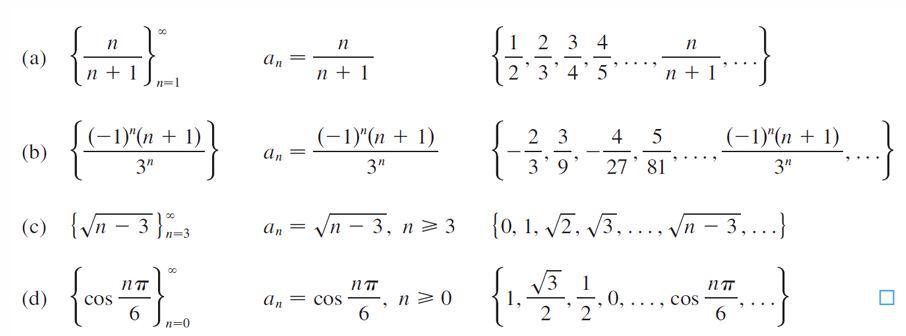用 k-NN 算法给企鹅 “分类”:从数据清洗到模型落地的完整指南
一、数据集初探:认识企鹅数据
首先导入常用的库,然后查看 seaborn 库中可用的数据集。本实验将使用企鹅(penguins)数据集
# 导入所需库
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets, linear_model
import pandas as pd
import seaborn as sns
# 查看seaborn库中的数据集名称
sns.get_dataset_names()
输出
['anagrams', 'anscombe', 'attention', 'brain_networks', 'car_crashes', 'diamonds', 'dots', 'dowjones', 'exercise', 'flights', 'fmri', 'geyser', 'glue', 'iris', 'healthexp', 'mpg', 'penguins', 'planets', 'seaice', 'taxis', 'tips', 'titanic']接下来,我们加载企鹅数据集并查看其内容。将数据集加载到一个名为 dfp(即 penguins dataframe,企鹅数据框)的数据框中,然后查看该数据表的前几行数据
# 加载企鹅数据集
dfp = sns.load_dataset('penguins')
# 查看数据集的前几行
dfp.head()
# 查看数据集的后几行
dfp.tail()
# 获取数据集的行数和列数
num_rows, num_columns = dfp.shape
# 打印行数(数据点/观测值数量)和列数(特征/测量指标数量)
print('数据点(或观测值)数量 =', num_rows) #数据点(或观测值)数量 = 344
print('特征(或测量指标)数量 =', num_columns)。#特征(或测量指标)数量 = 7还可以通过散点图来可视化数据:
# 绘制以体重为x轴、喙深度为y轴,按物种分类的散点图
sns.scatterplot(data=dfp, x="body_mass_g", y="bill_depth_mm", hue="species")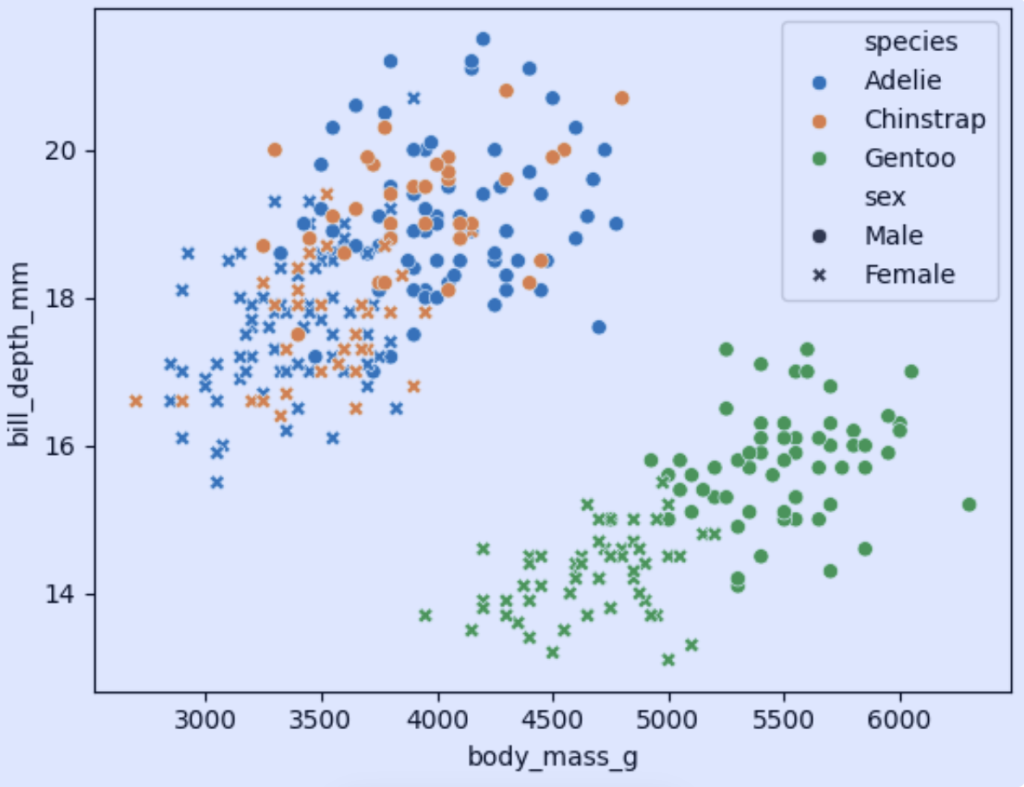
如果觉得上述散点图看起来有些拥挤,可以通过以下代码调整图形大小:
# 设置图形大小
plt.figure(figsize=(8, 8))
# 绘制以体重为x轴、喙深度为y轴,按岛屿分类的散点图
sns.scatterplot(data=dfp, x="body_mass_g", y="bill_depth_mm", hue="island")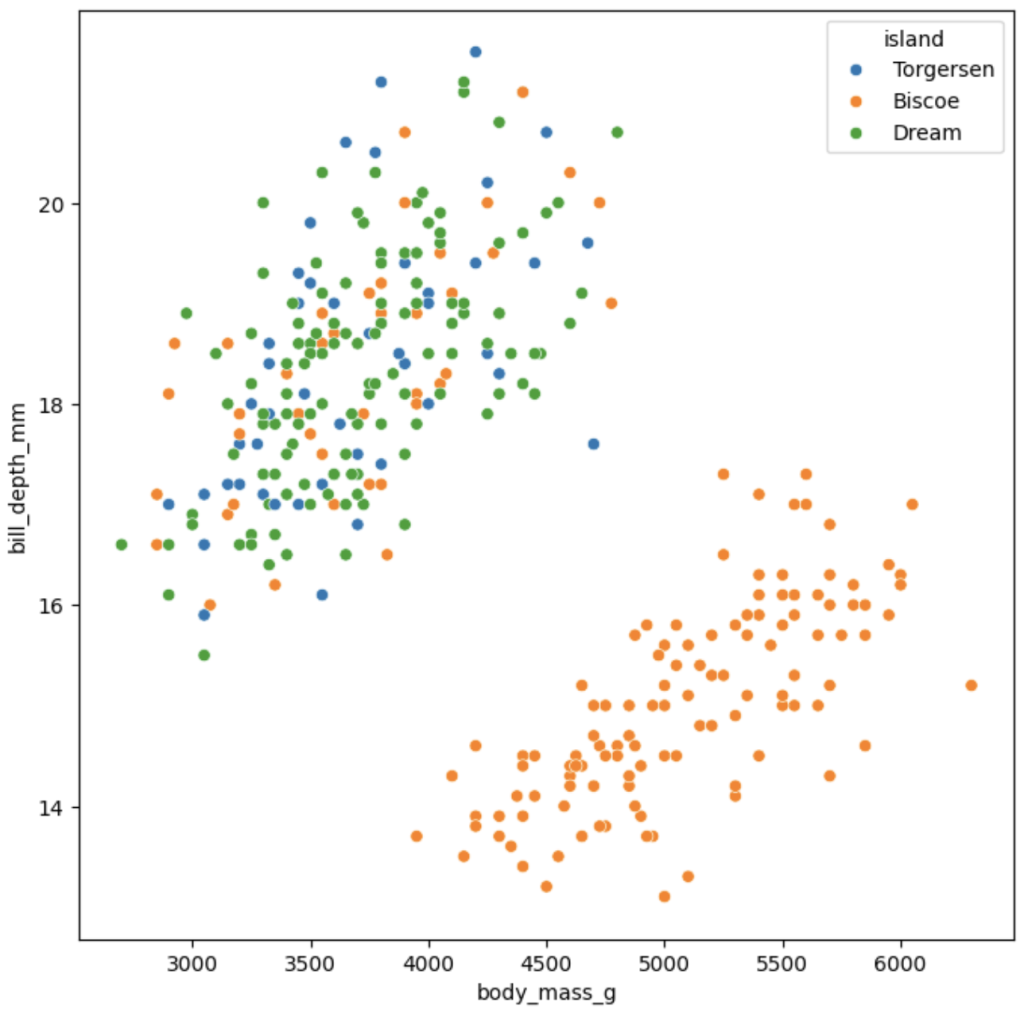
# 如果不通过可视化打印整个数据集(取消注释后执行该单元格,查看完后需重新注释以清除大量输出)
# print(dfp.to_string())
# 查看“species”列中的唯一值
dfp.species.unique()
# 也可以这样写,访问“species”列的数据
dfp2['species']
dfp2['species'].unique()# 数据集中存在一些无效值(NaN,即 “非数字”),统计每列中无效值(NaN)的数量
dfp.isna().sum()
#输出:
species 0
island 0
bill_length_mm 2
bill_depth_mm 2
flipper_length_mm 2
body_mass_g 2
sex 11
dtype: int64
# 找出包含无效值(NaN)的行
NaN_rows = dfp[dfp.isna().any(axis=1)]
# 查看这些包含无效值的行
NaN_rows
# 打印包含无效值的行的索引
print(NaN_rows.index)
//输出Index([3, 8, 9, 10, 11, 47, 246, 286, 324, 336, 339], dtype='int64')
# 通过索引查看包含无效值的行
dfp.loc[NaN_rows.index]dfp.isna()
isna() 是 Pandas 中用于检测缺失值的方法。它会遍历 dfp 这个 DataFrame 的每个元素,若元素是 NaN(或其他 Pandas 认定的缺失值形式,比如 None 等),就返回 True;否则返回 False。
执行后,会得到一个和原 dfp 形状完全相同 的布尔型 DataFrame(每个位置都是 True 或 False,标记对应位置是否为缺失值)。
any(axis = 1)
any() 是用于判断 “是否存在满足条件的元素” 的方法。
axis=1 表示 按 “行” 进行判断:对于每一行,只要这一行里有 任意一个 元素是 True(即原数据中该行存在 NaN),整个行就会被标记为 True;只有当一行里所有元素都是 False(即该行没有 NaN),才会标记为 False。
执行后,会得到一个布尔型的 Series(长度等于原 DataFrame 的行数),每个元素标记对应行是否包含 NaN。
处理缺失值
均值填充(Mean Imputation)
处理缺失值的一种方法是用 “合理” 的值填充这些缺失值。均值填充的实现方法简单(如下述仅用一行代码即可实现),但需要注意的是,这种方法会改变原始数据集。
均值填充的优点是可以保持样本数量不变;缺点是它可能会改变数据的某些统计特性(例如,数据的方差可能会发生变化)
下面以数值型特征用均值填充、缺失的性别(sex)特征均视为 “雌性(Female)” 为例进行处理:
# 对缺失值进行填充:数值型特征用均值填充,性别特征缺失值填充为'Female'
dfp1 = dfp.fillna({
'bill_length_mm': dfp['bill_length_mm'].mean(),
'bill_depth_mm': dfp['bill_depth_mm'].mean(),
'flipper_length_mm': dfp['flipper_length_mm'].mean(),
'body_mass_g': dfp['body_mass_g'].mean(),
'sex': 'Female'
})dfp.fillna
dfp 是 Pandas 的 DataFrame 类型
dfp.fillna(value=None, method=None, axis=None, inplace=False, limit=None, downcast=None)value:用于填充缺失值的具体值或字典 / Series/DataFrame
案例中只显示传入Value参数,传入了一个字典 {...} 作为 value 的值,用于指定不同列的缺失值填充规则
对性别特征(sex),用固定字符串 'Female' 填充缺失值。
对数值型特征(bill_length_mm、bill_depth_mm 等),用各列自身的均值(dfp['列名'].mean())填充缺失值;
查看处理结果
# 查看处理后的数据框中,原本包含缺失值的行的情况(缺失值已被填充)
dfp1.loc[NaN_rows.index]
# 查看处理前的数据框中,包含缺失值的行的情况(原始缺失值)
dfp.loc[NaN_rows.index]species(物种) | island(岛屿) | bill_length_mm(喙长度:毫米) | bill_depth_mm(喙深度:毫米) | flipper_length_mm(鳍长度:毫米) | body_mass_g(体重:克) | sex(性别) | |
|---|---|---|---|---|---|---|---|
| 3 | Adelie(阿德利企鹅) | Torgersen(托尔格森岛) | 43.92193 | 17.15117 | 200.915205 | 4201.754386 | Female(雌性) |
| 8 | Adelie(阿德利企鹅) | Torgersen(托尔格森岛) | 34.10000 | 18.10000 | 193.000000 | 3475.000000 | Female(雌性) |
| 9 | Adelie(阿德利企鹅) | Torgersen(托尔格森岛) | 42.00000 | 20.20000 | 190.000000 | 4250.000000 | Female(雌性) |
| 10 | Adelie(阿德利企鹅) | Torgersen(托尔格森岛) | 37.80000 | 17.10000 | 186.000000 | 3300.000000 | Female(雌性) |
| 11 | Adelie(阿德利企鹅) | Torgersen(托尔格森岛) | 37.80000 | 17.30000 | 180.000000 | 3700.000000 | Female(雌性) |
| 47 | Adelie(阿德利企鹅) | Dream(梦岛) | 37.50000 | 18.90000 | 179.000000 | 2975.000000 | Female(雌性) |
| 246 | Gentoo(巴布亚企鹅) | Biscoe(比斯科岛) | 44.50000 | 14.30000 | 216.000000 | 4100.000000 | Female(雌性) |
| 286 | Gentoo(巴布亚企鹅) | Biscoe(比斯科岛) | 46.20000 | 14.40000 | 214.000000 | 4650.000000 | Female(雌性) |
| 324 | Gentoo(巴布亚企鹅) | Biscoe(比斯科岛) | 47.30000 | 13.80000 | 216.000000 | 4725.000000 | Female(雌性) |
| 336 | Gentoo(巴布亚企鹅) | Biscoe(比斯科岛) | 44.50000 | 15.70000 | 217.000000 | 4875.000000 | Female(雌性) |
| 339 | Gentoo(巴布亚企鹅) | Biscoe(比斯科岛) | 43.92193 | 17.15117 | 200.915205 | 4201.754386 | Female(雌性) |
在数据处理过程中,检查处理结果是一种良好的习惯。但对于大型数据集而言,由于无法逐行打印并检查每个数据项以确保没有引入错误,因此需要采用其他有效的检查方法。
- 一种方法是绘制处理前后数据集的图形,观察图形是否基本一致
# 绘制处理前数据集的散点图(以体重为x轴,喙深度为y轴)
sns.scatterplot(data=dfp, x="body_mass_g", y="bill_depth_mm")- 此外,可以使用 describe () 函数查看数据的统计摘要,对比处理前后数据的统计特性是否大致相同
# 查看处理前数据集的统计摘要
dfp.describe()
# 查看处理后数据集的统计摘要
dfp1.describe()bill_length_mm(喙长度:毫米) | bill_depth_mm(喙深度:毫米) | flipper_length_mm(鳍长度:毫米) | body_mass_g(体重:克) | |
|---|---|---|---|---|
| count(数量) | 342.000000 | 342.000000 | 342.000000 | 342.000000 |
| mean(均值) | 43.921930 | 17.151170 | 200.915205 | 4201.754386 |
| std(标准差) | 5.459584 | 1.974793 | 14.061714 | 801.954536 |
| min(最小值) | 32.100000 | 13.100000 | 172.000000 | 2700.000000 |
| 25%(第一四分位数) | 39.225000 | 15.600000 | 190.000000 | 3550.000000 |
| 50%(中位数) | 44.450000 | 17.300000 | 197.000000 | 4050.000000 |
| 75%(第三四分位数) | 48.500000 | 18.700000 | 213.000000 | 4750.000000 |
| max(最大值) | 59.600000 | 21.500000 | 231.000000 | 6300.000000 |
删除包含空值的行
# 查看包含缺失值的行
dfp.loc[NaN_rows.index]
# 统计每列中缺失值(NaN)的数量
dfp.isna().sum()
# 删除包含缺失值的行,得到新的数据集dfp2
dfp2 = dfp.dropna()
#处理后的数据集 dfp2 中已不存在 NaN 值。统计dfp2中每列的缺失值数量,以验证这一点:
dfp2.isna().sum()# 查看删除缺失值后的数据集dfp2
dfp2注意到 dfp2 最左侧列的索引值不连续(例如,缺少索引 3)。我们可以使用 reset_index () 函数重置索引,但需要注意删除原始索引,否则原始索引会作为新列保留在数据框中:
# 错误的做法:这样会保留原始索引作为新列
# dfp2 = dfp2.reset_index()
# 正确的做法:重置索引并删除原始索引列
dfp2 = dfp2.reset_index(drop=True)
# 查看重置索引后的dfp2
dfp2数据可视化
接下来,我们将通过几种可视化方式来探索清理后的企鹅数据集
散点图
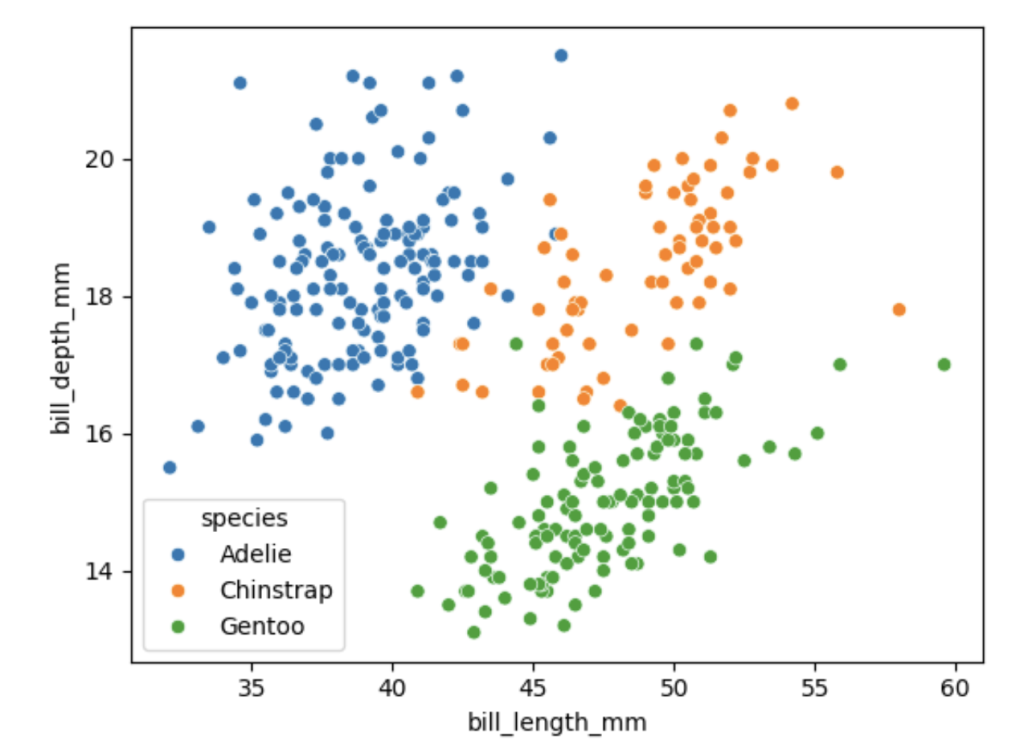
# 绘制以喙长度为x轴、喙深度为y轴,按物种分类的散点图
sns.scatterplot(data=dfp2, x="bill_length_mm", y="bill_depth_mm", hue="species")scatterplot
seaborn.scatterplot(data=None, x=None, y=None, hue=None, size=None,
style=None, alpha=None, s=None, ...)基础数据参数
data:传入的数据集(通常是 Pandas DataFrame),后续参数可直接使用列名。x/y:指定散点图的横轴和纵轴变量(必选,通常为数值型)。
分组与样式参数
hue:按指定变量对数据分组,并用不同颜色区分(如按 “性别” 分组,不同性别点的颜色不同)。size:按指定变量设置点的大小(如用 “年龄” 决定点的大小)。style:按指定变量设置点的形状(如按 “类别” 用圆形 / 三角形区分)。
外观参数
alpha:点的透明度(0~1,值越小越透明,避免点重叠时看不清)。s:点的固定大小(若不通过size动态设置,可直接指定固定值,如s=100)。
成对关系图
# 绘制按物种分类的成对关系图
sns.pairplot(dfp2, hue='species')pairplot
seaborn.pairplot(data, hue=None, hue_order=None, palette=None, vars=None, x_vars=None, y_vars=None, kind=’scatter’, diag_kind=’auto’, corner=False, …)
基础数据参数
vars:指定要分析的变量列表(默认使用所有数值型变量)。例如vars=['身高', '体重', '年龄']只分析这三个变量。data:必选,输入的数据集(通常是 Pandas DataFrame)。x_vars/y_vars:分别指定横轴和纵轴的变量(可用于绘制非对称的关系图)。
分组与样式参数
hue:按指定的类别变量分组,用不同颜色区分各组数据(如按 “性别” 分组,不同性别点的颜色不同)。palette:指定颜色方案(如palette='Set2')。
图表类型参数
kind:非对角线位置的图表类型,默认'scatter'(散点图),可选'reg'(添加回归线)、'kde'(核密度图,展示密度分布)。diag_kind:对角线位置的单变量分布图类型,默认'auto'(数值型变量用直方图),可选'hist'(直方图)、'kde'(核密度图)。
布局参数
corner:是否只绘制左下角的三角区域(避免重复,因为变量 A 与 B 的关系和 B 与 A 的关系相同),corner=True 可简化图表。
图中每个子图展示两个变量之间的关系: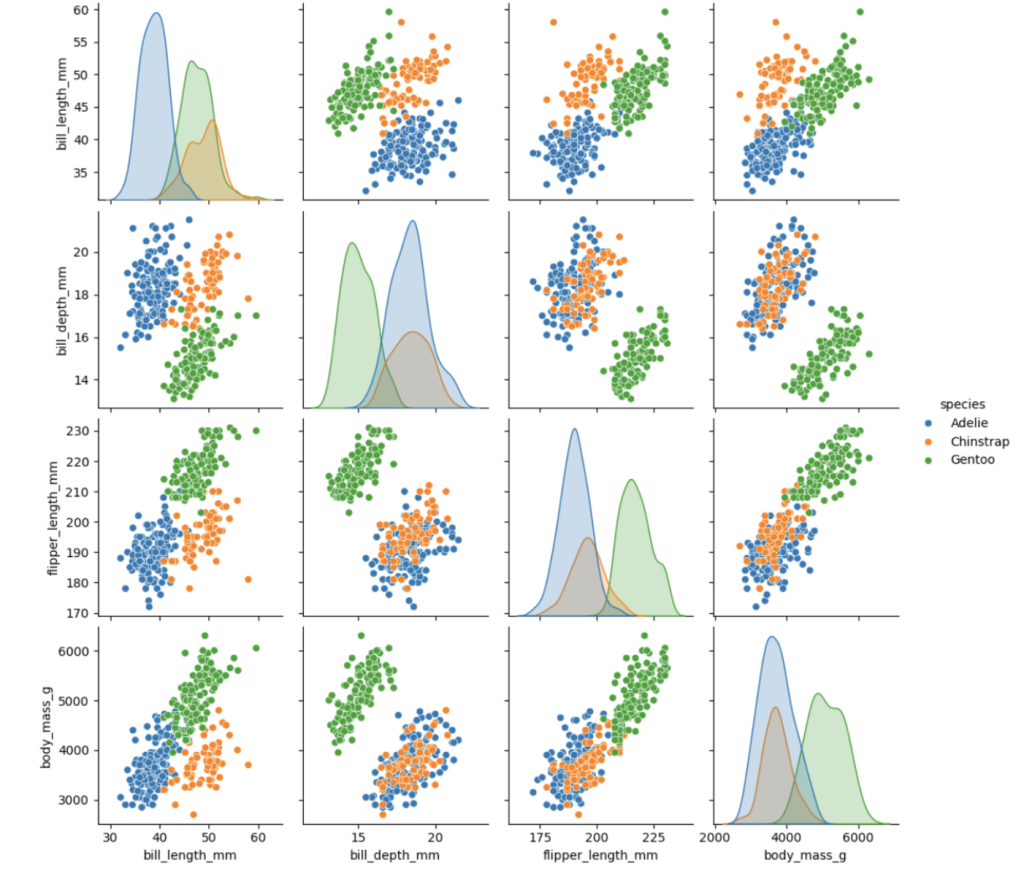
- 非对角线子图:是散点图,用于观察两个变量(如
bill_length_mm和bill_depth_mm)之间的相关性(正相关、负相关或无明显相关),还能通过颜色区分不同类别(这里是企鹅的不同物种species),看出不同类别在变量关系上的差异。 - 对角线子图:是单变量的分布直方图(或核密度图),展示单个变量(如
bill_length_mm)自身的分布特征(如是否对称、峰值位置等),同时不同颜色也能体现该变量在不同类别中的分布差异。
# 绘制按物种分类的角形成对关系图(仅显示下三角部分)
# 参考链接:https://seaborn.pydata.org/generated/seaborn.pairplot.html
sns.pairplot(dfp2, corner=True, hue='species', height=1.5)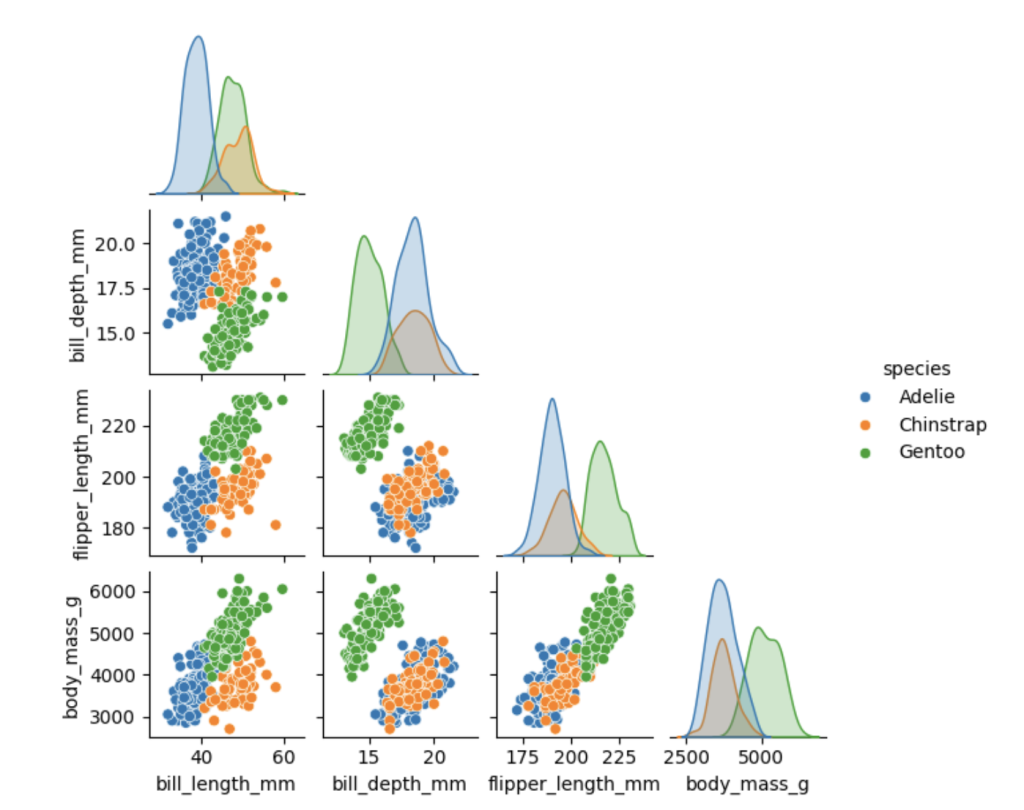
# 绘制按物种分类的成对关系图,对角线为核密度估计图,下三角添加核密度曲线
g = sns.pairplot(dfp2, diag_kind="kde", hue='plot(dfp2, diag_kind="kde", hue='species')
g.map_lower(sns.kdeplot, levels=4, color=".2")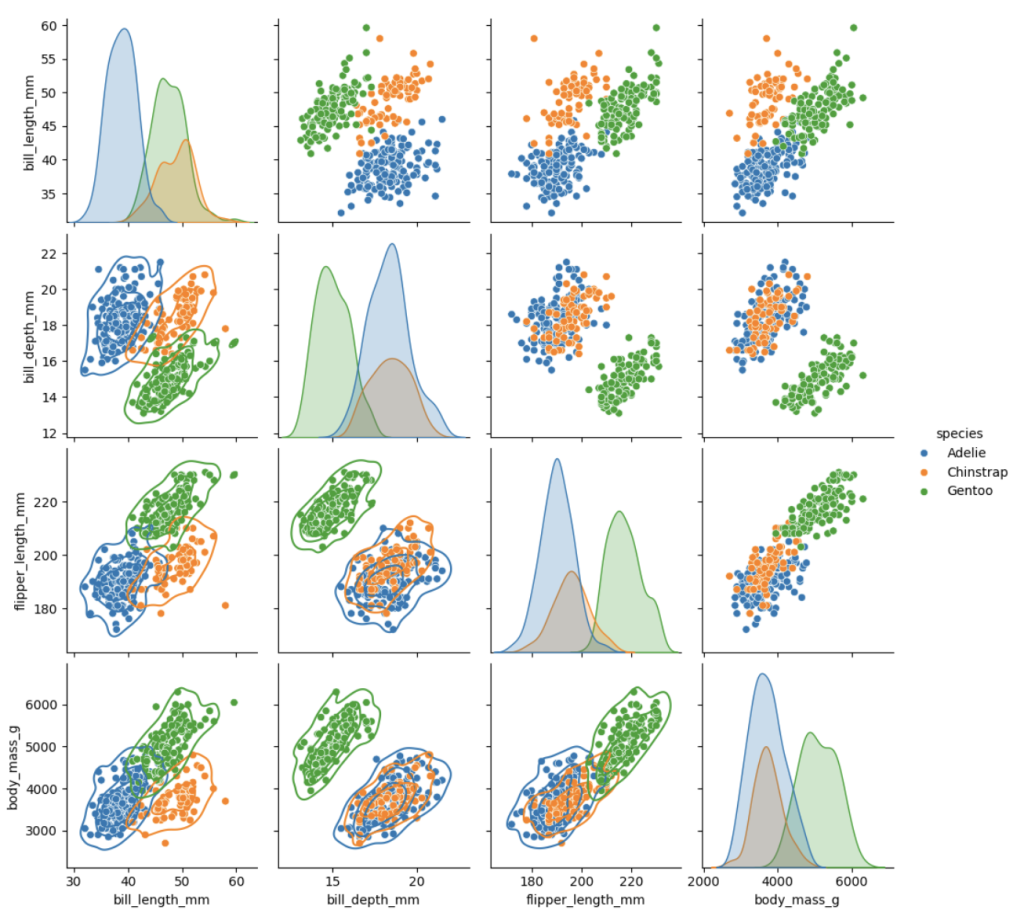
此处为一个成对关系图,对角线为各特征的核密度估计图,非对角线为不同特征之间的散点图,下三角部分还添加了 4 个级别的核密度曲线,不同物种的企鹅用不同颜色标注
创建数据子集
有时,根据某个特征值将数据集分离成不同的子集是很有用的。例如,如果我们选择按 “species”(物种)特征分离数据,可以使用以下命令获取仅包含阿德利企鹅(Adelie)数据的新数据框:
dfp2.loc[dfp2['species'] == 'Adelie']我们可以创建三个数据子集,分别对应三种企鹅物种:
# 创建阿德利企鹅(Adelie)的数据子集
dfA = dfp2.loc[dfp2['species'] == 'Adelie']
# 创建帽带企鹅(Chinstrap)的数据子集
dfC = dfp2.loc[dfp2['species'] == 'Chinstrap']
# 创建巴布亚企鹅(Gentoo)的数据子集
dfG = dfp2.loc[dfp2['species'] == 'Gentoo']该命令的工作原理是:dfp2['species'] == 'Adelie' 会对名为species列中的每一行进行判断,如果该行的 “species” 特征值为 “Adelie”,则返回 “True”;然后 dfp2.loc[?] 会保留所有返回 “True” 的行。我们可以将这些行分配给一个新的数据框。